Mediante el uso de metagenómica de tercera generación, investigadores del Departamento de Producción Vegetal y Microbiología de la Universidad Miguel Hernández de Elche (UMH) han logrado descifrar el éxito evolutivo de la bacteria marina SAR11, la más abundante del océano, desde un punto de vista genómico.
 El estudio revela que estas bacterias combinan un ‘núcleo genético’ común con pequeñas regiones de ‘genes flexibles’ que les permiten a nivel de población adaptarse con éxito a las fluctuaciones medioambientales . Este avance permitirá entender cómo estas poblaciones microbianas, claves para el equilibrio ecológico global, se diversifican y sobreviven al cambio climático.
El estudio revela que estas bacterias combinan un ‘núcleo genético’ común con pequeñas regiones de ‘genes flexibles’ que les permiten a nivel de población adaptarse con éxito a las fluctuaciones medioambientales . Este avance permitirá entender cómo estas poblaciones microbianas, claves para el equilibrio ecológico global, se diversifican y sobreviven al cambio climático.
El microbioma marino desempeña un papel esencial en el mantenimiento de los ecosistemas, impulsando los ciclos biogeoquímicos globales y representando hasta el 98% de la productividad primaria marina. El grupo de bacterias denominado clado SAR11 son de vida libre y dominan numéricamente las aguas superficiales del océano, representando el 20-40% de todas las células procariotas. “A pesar de la abundancia y distribución cosmopolita de estos microbios, las limitaciones para recuperar toda la riqueza genética de sus poblaciones naturales han impedido desentrañar la relación entre evolución y ecología microbiana desde un punto de vista genómico.” apunta el investigador de la UMH y líder del estudio Mario López Pérez.
El Grupo de Genómica y Evolución Microbiana de la UMH ha combinado por primera vez técnicas de genómica de célula única y metagenómica de lectura larga para reconstruir con alta precisión la diversidad genética de SAR11 en muestras ambientales del Mediterráneo. Esta combinación ha permitido descifrar cómo se organiza su genoma y cómo se diversifican las cepas que conviven en una misma población.
 El estudio muestra que estas bacterias comparten un núcleo genético casi idéntico, que representa el 81% de su genoma. El resto, conocido como genoma flexible, está concentrado en pequeñas regiones —la mayoría con un único gen— y se encuentra en posiciones equivalentes dentro de todas las cepas. “Estas pequeñas variaciones están siempre en el mismo lugar del genoma y contienen genes con funciones equivalentes, aunque en distintas versiones”, explica la investigadora de la UMH Carmen Molina Pardines, primera firmante del estudio. Este patrón genómico favorece la coexistencia de múltiples cepas y reduce la competencia entre ellas.
El estudio muestra que estas bacterias comparten un núcleo genético casi idéntico, que representa el 81% de su genoma. El resto, conocido como genoma flexible, está concentrado en pequeñas regiones —la mayoría con un único gen— y se encuentra en posiciones equivalentes dentro de todas las cepas. “Estas pequeñas variaciones están siempre en el mismo lugar del genoma y contienen genes con funciones equivalentes, aunque en distintas versiones”, explica la investigadora de la UMH Carmen Molina Pardines, primera firmante del estudio. Este patrón genómico favorece la coexistencia de múltiples cepas y reduce la competencia entre ellas.
El estudio revela que estas bacterias forman poblaciones policlonales, es decir, compuestas por múltiples variantes genéticas que conviven en el mismo entorno. Este proceso no sólo garantiza la conservación de los genes esenciales durante los barridos selectivos, sino que también mantiene la redundancia funcional, salvaguardando una amplia reserva genética ambiental. Este reservorio proporciona a la población la capacidad de dar respuestas rápidas y adaptativas a las fluctuaciones medioambientales. “Estos resultados nos dan pistas sobre las estrategias que explican el éxito ecológico de SAR11 en ambientes marinos pobres en nutrientes, como el Mediterráneo”, aclara el investigador de la UMH José M. Haro Moreno, también primer firmante.
Más allá del descubrimiento evolutivo, el estudio demuestra que la metagenómica de tercera generación permite superar las limitaciones técnicas que impedían estudiar estos microorganismos que son extremadamente difíciles de obtener en cultivo puro.
El estudio, publicado en la revista científica Microbiome, posiciona a la UMH como referente internacional en el estudio evolutivo del microbioma marino. Su enfoque integrador permite entender mejor los mecanismos que sostienen la vida en los océanos y, por extensión, en el planeta.
Este trabajo ha sido financiado por los proyectos “FLEX3GEN” y “MICRO3GEN”, del Ministerio de Economía, Industria y Competitividad, con cofinanciación de fondos FEDER. Además, la investigación ha contado con el apoyo de becas de doctorado del Ministerio (para Carmen Molina Pardines) y del programa Margarita Salas, cofinanciado por el Ministerio de Universidades y la Unión Europea – Next Generation EU (para José M. Haro Moreno).
Acceso al artículo: Molina-Pardines, C., Haro-Moreno, J. M., Rodriguez-Valera, F., & López-Pérez, M. (2025). Extensive paralogism in the environmental pangenome: a key factor in the ecological success of natural SAR11 populations. Microbiome, 13(1), 41. https://doi.org/10.1186/s40168-025-02037-6
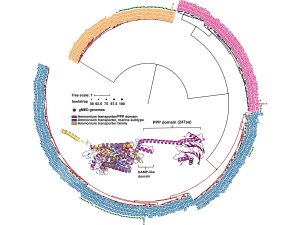
Pie de foto: En la imagen se muestra el ‘árbol genealógico’ de un grupo de genes llamados Amt, presentes en las bacterias SAR11. Las distintas versiones de estos genes —que pueden influir en cómo la bacteria capta y procesa el nitrógeno— se representan con colores diferentes. Las ramas marrones indican las versiones comunes a toda la población, mientras que los puntos verdes marcan las variantes halladas en cepas del Mediterráneo. Las estructuras de estas proteínas también se muestran superpuestas, destacando que, aunque haya diferencias, todas conservan funciones similares. Esta variación genética es clave para que estas bacterias se adapten a distintos entornos marinos.
Fuente: Molina-Pardines et al., 2025, Microbiome. https://doi.org/10.1186/s40168-025-02037-6